Dowiedz się, jak używać narzędzi OCR, Apache Spark i innych komponentów Apache Hadoop do przetwarzania obrazów PDF na dużą skalę.
Technologie optycznego rozpoznawania znaków (OCR) znacznie się rozwinęły w ciągu ostatnich 20 lat. Jednak w tym czasie niewiele lub wcale nie było wysiłku, aby połączyć OCR z architekturą rozproszoną, taką jak Apache Hadoop, w celu przetwarzania dużej liczby obrazów w czasie zbliżonym do rzeczywistego.
W tym poście dowiesz się, jak używać standardowych narzędzi open source wraz z komponentami Hadoop, takimi jak Apache Spark, Apache Solr i Apache HBase, aby zrobić to w przypadku użycia informacji o urządzeniu medycznym. W szczególności użyjesz publicznego zbioru danych do konwersji tekstu narracyjnego na pola z możliwością wyszukiwania.
Chociaż ten przykład koncentruje się na informacjach o urządzeniu medycznym, można go zastosować w wielu innych scenariuszach, w których wymagane jest przetwarzanie i utrwalanie obrazów. Firmy ubezpieczeniowe mogą na przykład udostępnić wszystkie zeskanowane dokumenty w plikach roszczeń w celu lepszego rozstrzygnięcia roszczenia. Podobnie dział łańcucha dostaw w zakładzie produkcyjnym może skanować wszystkie arkusze danych technicznych od dostawców części i umożliwiać ich przeszukiwanie przez analityków.
Przypadek użycia:rejestracja urządzenia medycznego
W ostatnich latach nastąpiła lawina zmian w dziedzinie elektronicznej rejestracji produktów leczniczych. Standard ISO IDMP (identyfikacja produktów medycznych) jest jednym z takich formatów wiadomości do rejestracji produktów i zawartych w nich substancji, przy czym identyfikator produktu leczniczego, identyfikator opakowania i identyfikator partii są używane do śledzenia produktów w przypadku działań niepożądanych, nielegalnych import, podrabianie i inne kwestie związane z nadzorem nad bezpieczeństwem farmakoterapii. Norma wymaga, aby nie tylko nowe produkty musiały być rejestrowane, ale również starsze/zarchiwizowane dokumenty dotyczące każdego produktu, z którym opinia publiczna może być narażona, muszą być również dostarczane w formie elektronicznej.
Aby zachować zgodność ze standardami IDMP w różnych firmach, firmy muszą mieć możliwość pobierania i przetwarzania danych z wielu źródeł danych, takich jak RDBMS, a także, w niektórych przypadkach, ze starszych arkuszy danych produktów. Chociaż dobrze wiadomo, jak pozyskiwać dane z RDBMS za pomocą technologii takich jak Apache Sqoop, przetwarzanie starszych dokumentów wymaga nieco więcej pracy. W większości dokumenty muszą zostać przetworzone, a odpowiedni tekst musi być programowo wyodrębniony na dużą skalę przy użyciu istniejących technologii OCR.
Zbiór danych
Użyjemy zbioru danych z FDA, który zawiera wszystkie zgłoszenia 510(k) kiedykolwiek złożone przez producentów urządzeń medycznych od 1976 roku. Sekcja 510(k) ustawy o żywności, lekach i kosmetykach wymaga od producentów urządzeń, którzy muszą się zarejestrować, aby powiadomić FDA o zamiarze wprowadzenia na rynek urządzenia medycznego z co najmniej 90-dniowym wyprzedzeniem.
Ten zbiór danych jest przydatny w tym przypadku z kilku powodów:
- Dane są bezpłatne i należą do domeny publicznej.
- Dane są zgodne z rozporządzeniem europejskim, które wchodzi w życie w lipcu 2016 r. (gdzie producenci muszą przestrzegać nowych standardów danych). Wypełnienia FDA zawierają ważne informacje istotne dla uzyskania pełnego obrazu IDMP.
- Format dokumentów (PDF) pozwala nam zademonstrować proste, ale skuteczne techniki OCR w przypadku dokumentów w wielu formatach.
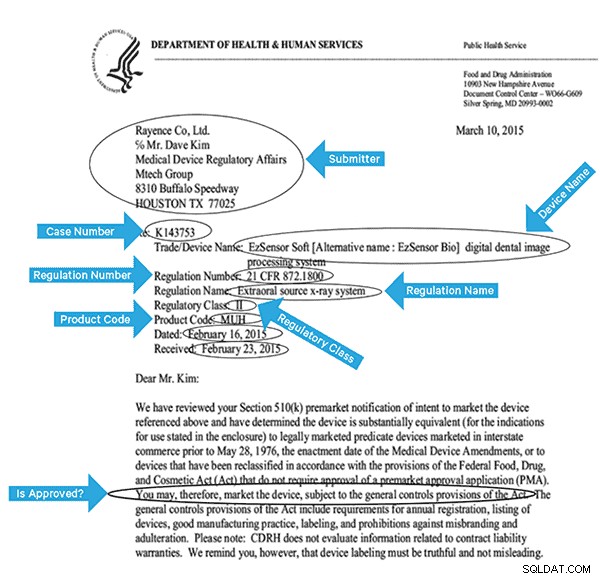
Aby skutecznie zindeksować te dane, musimy wyodrębnić niektóre pola z obrazów. Poniżej znajduje się przykładowy dokument z potencjalnymi polami, które można wyodrębnić.
Architektura wysokiego poziomu
W tym przypadku pliki PDF są przechowywane w HDFS i przetwarzane przy użyciu bibliotek Spark i OCR. (Krok przetwarzania wykracza poza zakres tego posta, ale może być tak prosty, jak uruchomienie hdfs -dfs -put lub przy użyciu interfejsu webhdfs). nowa funkcjonalność MOB, do rozruchu. Cloudera Search (zbudowany na bazie Apache Solr) to jedyne rozwiązanie do wyszukiwania, które integruje się natywnie z HBase, umożliwiając w ten sposób tworzenie indeksów pomocniczych.
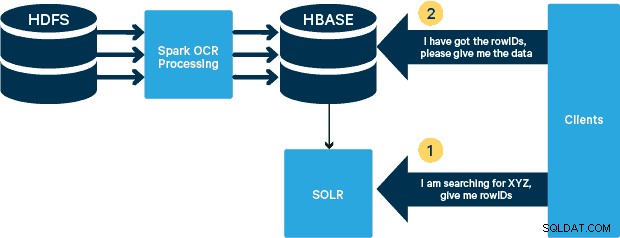
Konfigurowanie tabeli urządzeń medycznych w HBase
Zachowamy prosty schemat dla naszego przypadku użycia. RowID będzie nazwą pliku i będą istniały dwie rodziny kolumn:„info” i „obj”. Rodzina kolumn „info” będzie zawierać wszystkie pola, które wyodrębniliśmy z obrazów. Rodzina kolumn „obj” będzie zawierać bajty rzeczywistego obiektu binarnego, w tym przypadku PDF. Nazwa tabeli w naszym przypadku to „mdds”.
Skorzystamy z funkcjonalności HBase MOB (medium object) wprowadzonej w HBASE-11339. Aby skonfigurować HBase do obsługi MOB, wymagane jest kilka dodatkowych kroków, ale wygodnie instrukcje można znaleźć pod tym linkiem.
Istnieje wiele sposobów programowego tworzenia tabeli w HBase (Java API, REST API lub podobna metoda). Tutaj użyjemy powłoki HBase do utworzenia tabeli „mdds” (celowo przy użyciu opisowej nazwy rodziny kolumn, aby ułatwić śledzenie). Chcemy, aby rodzina kolumn „informacje” została zreplikowana do Solr, ale nie dane MOB.
Poniższe polecenie utworzy tabelę i włączy replikację w rodzinie kolumn o nazwie „informacje”. Bardzo ważne jest określenie opcji REPLICATION_SCOPE => '1' , w przeciwnym razie HBase Lily Indexer nie otrzyma żadnych aktualizacji z HBase. Chcemy użyć ścieżki MOB w HBase dla obiektów większych niż 10 MB. Aby to osiągnąć, tworzymy również inną rodzinę kolumn, zwaną „obj”, używając następujących parametrów dla obiektów MOB:
IS_MOB => true, MOB_THRESHOLD => 10240000
IS_MOB parametr określa, czy ta rodzina kolumn może przechowywać obiekty MOB, podczas gdy MOB_THRESHOLD określa, jak duży musi być obiekt, aby można go było uznać za MOB. Stwórzmy więc tabelę:
create 'mdds', {NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF',REPLICATION_SCOPE => '1'},{NAME => 'obj', IS_MOB => true, MOB_THRESHOLD => 10240000}
Aby potwierdzić, że tabela została utworzona poprawnie, uruchom następujące polecenie w powłoce HBase:
hbase(main):001:0> describe 'mdds'
Table mdds is ENABLED
mdds
COLUMN FAMILIES DESCRIPTION
{NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '1', VERSIONS => '1', COMPRESSION => 'NONE', MIN_VERSIONS => '0', TTL => 'FOREVER', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}
{NAME => 'obj', DATA_BLOCK_ENCODING => 'NONE', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '0', COMPRESSION => 'NONE', VERSIONS => '1', MIN_VERSIONS => '0', TTL => 'FOREVER', MOB_THRESHOLD => '10240000', IS_MOB => 'true', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}
2 row(s) in 0.3440 seconds
Przetwarzanie zeskanowanych obrazów za pomocą Tesseract
OCR przeszedł długą drogę, jeśli chodzi o radzenie sobie z odmianami czcionek, szumem obrazu i problemami z wyrównaniem. Tutaj użyjemy otwartego silnika OCR Tesseract, który został pierwotnie opracowany jako zastrzeżone oprogramowanie w laboratoriach HP. Rozwój Tesseract został od tego czasu wydany jako oprogramowanie typu open source i od 2006 roku jest sponsorowany przez Google.
Tesseract to wysoce przenośna biblioteka oprogramowania. Wykorzystuje bibliotekę przetwarzania obrazu Leptonica do generowania obrazu binarnego poprzez adaptacyjne progowanie na szarym lub kolorowym obrazie.
Przetwarzanie odbywa się według tradycyjnego procesu krok po kroku. Poniżej przedstawiono przybliżony przebieg kroków:
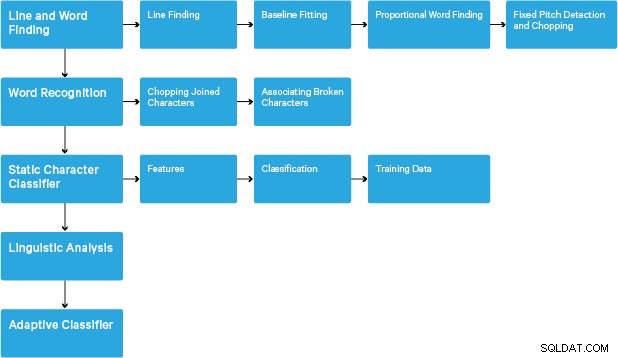
Przetwarzanie rozpoczyna się od analizy połączonych komponentów, co skutkuje przechowywaniem znalezionych komponentów. Ten krok pomaga w sprawdzeniu zagnieżdżenia konturów oraz liczby konturów dzieci i wnuków.
Na tym etapie kontury są zbierane razem, wyłącznie przez zagnieżdżanie, w dużych obiektach binarnych (BLOB). Bloki BLOB są zorganizowane w linie tekstu, a linie i regiony są analizowane pod kątem tekstu o stałej gęstości lub tekstu proporcjonalnego. Linie tekstu są różnie dzielone na słowa w zależności od rodzaju odstępów między znakami. Tekst o stałej wysokości jest natychmiast posiekany przez komórki znaków. Tekst proporcjonalny jest dzielony na słowa przy użyciu spacji określonych i spacji rozmytych.
Rozpoznawanie przebiega wtedy jako proces dwuprzebiegowy. W pierwszym przejściu próbuje się kolejno rozpoznawać każde słowo. Każde zadowalające słowo jest przekazywane do klasyfikatora adaptacyjnego jako dane treningowe. Klasyfikator adaptacyjny ma wtedy szansę na dokładniejsze rozpoznanie tekstu na dole strony. Ponieważ klasyfikator adaptacyjny mógł nauczyć się czegoś przydatnego zbyt późno, aby wnieść wkład w górnej części strony, drugi przebieg jest przeprowadzany po stronie, w którym słowa, które nie zostały rozpoznane wystarczająco dobrze, są ponownie rozpoznawane. Ostatnia faza rozwiązuje spacje rozmyte i sprawdza alternatywne hipotezy dotyczące wysokości x, aby zlokalizować tekst kapitalikami.
Tesseract w swojej obecnej formie jest w pełni zdolny do Unicode i jest wyszkolony w kilku językach. Z naszych badań wynika, że jest to jedna z najdokładniejszych bibliotek open source dostępnych dla OCR. Jak wspomniano wcześniej, Tesseract używa Leptoniki. Używamy również Ghostscript do dzielenia plików PDF na obrazy. (Możesz podzielić na dowolny format kompresji obrazu; wybraliśmy PNG). Te trzy biblioteki są napisane w C++ i aby wywoływać je z programów Java/Scala, musimy użyć implementacji odpowiednich interfejsów Java Native. W naszej pracy wykorzystujemy powiązania JNI z JavaPresets. (Instrukcje kompilacji można znaleźć poniżej.) Użyliśmy Scali do napisania sterownika Spark.
val renderer :SimpleRenderer = new SimpleRenderer( ) renderer.setResolution( 300 ) val images:List[Image] = renderer.render( document )
Leptonica odczytuje podzielone obrazy z poprzedniego kroku.
ImageIO.write(
x.asInstanceOf[RenderedImage],
"png",
imageByteStream
)
val pix: PIX = pixReadMem (
ByteBuffer.wrap( imageByteStream.toByteArray( ) ).array( ),
ByteBuffer.wrap( imageByteStream.toByteArray( ) ).capacity( )
) Następnie używamy wywołań API Tesseract, aby wyodrębnić tekst. Zakładamy, że dokumenty są tutaj w języku angielskim, dlatego drugim parametrem metody Init jest „eng”.
val api: TessBaseAPI = new TessBaseAPI( ) api.Init( null, "eng" ) api.SetImage(pix) api.GetUTF8Text().getString()
Po przetworzeniu obrazów wyodrębniamy niektóre pola z tekstu i wysyłamy je do HBase.
def populateHbase (
fileName:String,
lines: String,
pdf:org.apache.spark.input.PortableDataStream) : Unit =
{
/** Configure and open a HBase connection */
val mddsTbl = _conn.getTable( TableName.valueOf( "mdds" ));
val cf = "info"
val put = new Put( Bytes.toBytes( fileName ))
/**
* Extract Fields here using Regexes
* Create Put objects and send to HBase
*/
val aAndCP = """(?s)(?m).*\d\d\d\d\d-\d\d\d\d(.*)\nRe: (\w\d\d\d\d\d\d).*""".r
……..
lines match {
case
aAndCP( addr, casenum ) => put.add( Bytes.toBytes( cf ),
Bytes.toBytes( "submitter_info" ),
Bytes.toBytes( addr ) ).add( Bytes.toBytes( cf ),
Bytes.toBytes( "case_num" ), Bytes.toBytes( casenum ))
case _ => println( "did not match a regex" )
}
…….
lines.split("\n").foreach {
val regNumRegex = """Regulation Number:\s+(.+)""".r
val regNameRegex = """Regulation Name:\s+(.+)""".r
……..
…….
_ match {
case regNumRegex( regNum ) => put.add( Bytes.toBytes( cf ),
Bytes.toBytes( "reg_num" ),
…….
…..
case _ => print( "" )
}
}
put.add( Bytes.toBytes( cf ), Bytes.toBytes( "text" ), Bytes.toBytes( lines ))
val pdfBytes = pdf.toArray.clone
put.add(Bytes.toBytes( "obj" ), Bytes.toBytes( "pdf" ), pdfBytes )
mddsTbl.put( put )
…….
}
Jeśli przyjrzysz się uważnie powyższemu kodowi, tuż przed wysłaniem obiektu Put do HBase, wstawiamy surowe bajty PDF do rodziny kolumn „obj” tabeli. Używamy HBase jako warstwy przechowywania wyodrębnionych pól, a także surowego obrazu. Dzięki temu aplikacja może w razie potrzeby szybko i wygodnie wyodrębnić oryginalny obraz. Pełny kod można znaleźć tutaj. (Warto zauważyć, że chociaż używaliśmy standardowych interfejsów API HBase do tworzenia obiektów Put dla HBase, w rzeczywistym systemie produkcyjnym, rozsądnie byłoby rozważyć użycie interfejsów API SparkOnHBase, które umożliwiają aktualizacje wsadowe do HBase z Spark RDD).
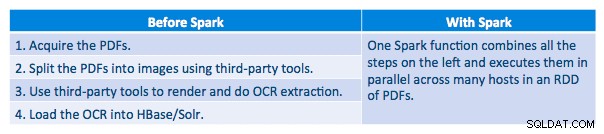
Potok wykonania
Udało nam się przetworzyć każdy plik PDF w seryjnym frameworku. Aby skalować przetwarzanie, zdecydowaliśmy się przetwarzać te pliki PDF w sposób rozproszony za pomocą Sparka. Poniższy wykres pokazuje, w jaki sposób łączymy różne etapy tego przetwarzania, aby przekształcić przepływ pracy w proste wywołanie makra ze Sparka i pobrać dane do HBase.
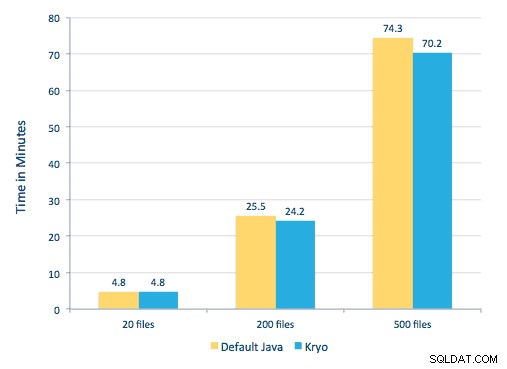
Próbowaliśmy również porównać metody serializacji, ale w naszym zestawie danych nie zauważyliśmy znaczącej różnicy w wydajności.
Konfiguracja środowiska
Użyty sprzęt:klaster z pięcioma węzłami z pamięcią 15 GB, 4 procesorami wirtualnymi i 2 dyskami SSD o pojemności 40 GB
Ponieważ do przetwarzania używaliśmy bibliotek C++, użyliśmy powiązań JNI, które można znaleźć tutaj.
Zbuduj powiązania JNI dla Tesseract i Leptonica z gotowych ustawień javaCPP:
-
- Na wszystkich węzłach:
yum -y install automake autoconf libtool zlib-devel libjpeg-devel giflib libtiff-devel libwebp libwebp-devel libicu-devel openjpeg-devel cairo-devel git clone https://github.com/bytedeco/javacpp-presets.gitcd javacpp-presets- Zbuduj Leptonica.
cd leptonica ./cppbuild.sh install leptonica cd cppbuild/linux-x86_64/leptonica-1.72/ LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configure make && sudo make install cd ../../../ mvn clean install cd ..
- Zbuduj Tesserakt.
- Na wszystkich węzłach:
cd tesseract ./cppbuild.sh install tesseract cd tesseract/cppbuild/linux-x86_64/tesseract-3.03 LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configure make && make install cd ../../../ mvn clean install cd ..
- Buduj ustawienia wstępne javaCPP.
mvn clean install --projects leptonica,tesseract
Używamy Ghostscript do wyodrębniania obrazów z plików PDF. Instrukcje budowania Ghostscript, odpowiadające użytym tutaj wersjom Tesseractu i Leptoniki, są następujące. (Upewnij się, że Ghostscript nie jest zainstalowany w systemie przez menedżera pakietów.)
wget https://downloads.ghostscript.com/public/ghostscript-9.16.tar.gz tar zxvf ghostscript-9.16.tar.gz cd ghostscript-9.16 ./autogen.sh && ./configure --prefix=/usr --disable-compile-inits --enable-dynamic sudo make && make soinstall && install -v -m644 base/*.h /usr/include/ghostscript && ln -v -s ghostscript /usr/include/ps (Depending on your ldpath setting, you may have to do) : sudo ln -sf /usr/lib/libgs.so /usr/local/lib/libgs.so
Upewnij się, że wszystkie potrzebne biblioteki znajdują się w ścieżce klasy. Wszystkie odpowiednie słoiki umieszczamy w katalogu o nazwie lib. Przecinek jest ważny poniżej:
$ for i in `ls lib/*`; do export MY_JARS=./$i,$MY_JARS; done tesseract.jar, tesseract-linux-x86_64.jar, javacpp.jar, ghost4j-1.0.0.jar, leptonica.jar, leptonica-1.72-1.0.jar, leptonica-linux-x86_64.jar
Wywołujemy program Spark w następujący sposób. Musimy określić extraLibraryPath dla natywnych bibliotek Ghostscript; druga konfiguracja jest potrzebna dla Tesseractu.
spark-submit --jars $MY_JARS --num-executors 12 --executor-memory 4G --executor- cores 1 --conf spark.executor.extraLibraryPath=/usr/local/lib --conf spark.executorEnv.TESSDATA_PREFIX=/home/vsingh/javacpp- presets/tesseract/cppbuild/1-x86_64/share/tessdata/ --conf spark.executor.extraClassPath=/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/ lib/htrace-core-3.1.0-incubating.jar --driver-class-path /etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/lib/htrace-core-3.1.0- incubating.jar --conf spark.serializer=org.apache.spark.serializer.KryoSerializer --conf spark.kryoserializer.buffer.mb=24 --class com.cloudera.sa.OCR.IdmpExtraction
Tworzenie kolekcji Solr
Solr dość bezproblemowo integruje się z HBase poprzez Lily HBase Indexer. Aby zrozumieć, w jaki sposób odbywa się integracja Lily Indexer z HBase, możesz odświeżyć nasz poprzedni post w sekcji „Zrozumienie replikacji HBase i Lily HBase Indexer”.
Poniżej przedstawiamy kroki, które należy wykonać, aby utworzyć indeksy:
- Wygeneruj przykładowy plik konfiguracyjny schema.xml:
solrctl --zk localhost:2181 instancedir --generate $HOME/solrcfg - Edytuj plik schema.xml w
$HOME/solrcfg, określając pola, których potrzebujemy do naszej kolekcji. Pełny plik można znaleźć tutaj. - Prześlij konfiguracje Solr do ZooKeepera:
solrctl --zk localhost:2181/solr instancedir --create mdds_collection $HOME/solrcfg - Wygeneruj kolekcję Solr z 2 fragmentami (-s 2) i 2 replikami (-r 2):
solrctl --zk localhost:2181/solr --solr localhost:8983/solr collection --create mdds_collection -s 2 -r 2
W powyższym poleceniu stworzyliśmy kolekcję Solr z dwoma parametrami shardów (-s 2) i dwoma replikami (-r 2). Parametry były wystarczające dla naszego korpusu, ale w rzeczywistym wdrożeniu należałoby ustawić liczbę w oparciu o inne względy wykraczające poza nasz zakres dyskusji tutaj.
Rejestracja indeksatora
Ten krok jest potrzebny do dodania i skonfigurowania indeksatora i replikacji HBase. Poniższe polecenie zaktualizuje ZooKeeper i doda mdds_indexer jako element równorzędny replikacji dla HBase. Wstawi również konfiguracje do ZooKeepera, których Lily HBase Indexer użyje do wskazania właściwej kolekcji w Solr. |
hbase-indexer add-indexer -n mdds_indexer -c indexer-config.xml -cp solr.zk=localhost:2181/solr -cp solr.collection=mdds_collection.
Argumenty:
-n mdds_indexer– określa nazwę indeksatora, który zostanie zarejestrowany w ZooKeeper-c indexer-config.xml– plik konfiguracyjny, który określi zachowanie indeksatora-cp solr.zk=localhost:2181/solr– określa lokalizację konfiguracji ZooKeeper i Solr. Powinno to zostać zaktualizowane o lokalizację ZooKeeper specyficzną dla środowiska.-cp solr.collection=mdds_collection– określa kolekcję do aktualizacji. Przypomnij sobie krok Konfiguracja Solr, w którym utworzyliśmy kolekcję1.
index-config.xml plik jest w tym przypadku stosunkowo prosty; wszystko, co robi, to określa indeksatorowi, na którą tabelę ma patrzeć, klasę, która będzie używana jako maper (com.ngdata.hbaseindexer.morphline.MorphlineResultToSolrMapper ) oraz lokalizację pliku konfiguracyjnego Morphline. Domyślnie typ mapowania jest ustawiony na wiersz , w takim przypadku dokument Solr staje się pełnym wierszem. Param name="morphlineFile" określa lokalizację pliku konfiguracyjnego Morphlines. Lokalizacja może być ścieżką bezwzględną pliku Morphlines, ale ponieważ używasz Cloudera Manager, określ ścieżkę względną jako morphlines.conf.
Zawartość pliku konfiguracyjnego hbase-indexer można znaleźć tutaj.
Konfigurowanie i uruchamianie Lily HBase Indexer
Po włączeniu Lily HBase Indexer należy określić logikę transformacji Morphlines, która pozwoli temu indeksatorowi analizować aktualizacje tabeli Medical Device i wyodrębniać wszystkie odpowiednie pola. Przejdź do Usługi i wybierz Lily HBase Indexer, który dodałeś wcześniej. Wybierz Konfiguracje->Wyświetl i edytuj->Usługa->Morphlines . Skopiuj i wklej plik Morphlines.
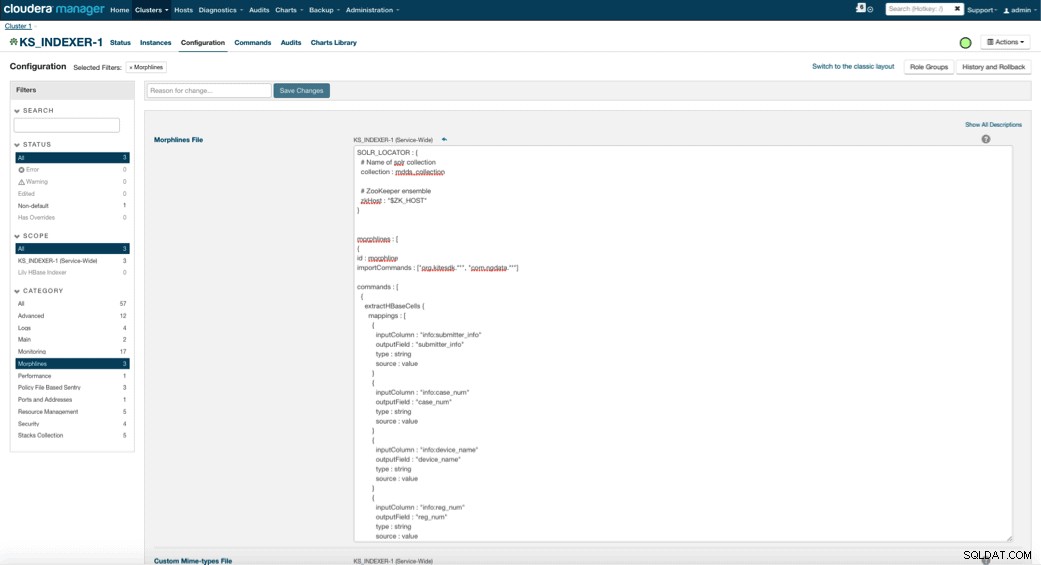
Biblioteka urządzeń medycznych morphlines wykona następujące działania:
- Odczytaj zdarzenia e-mail HBase za pomocą
extractHBaseCellspolecenie - Konwertuj datę/sygnatury czasowe na pole, które Solr zrozumie, za pomocą
convertTimestamppolecenia - Upuść wszystkie dodatkowe pola, których nie określiliśmy w schema.xml, za pomocą
sanitizeUknownSolrFieldspolecenie
Pobierz kopię tego pliku Morphlines stąd.
Ważną informacją jest to, że pole id zostanie automatycznie wygenerowane przez Lily HBase Indexer. To ustawienie można skonfigurować w powyższym pliku index-config.xml, określając atrybut pola klucza unikalnego. Najlepszą praktyką jest pozostawienie domyślnej nazwy identyfikatora — ponieważ nie została ona określona w powyższym pliku xml, wygenerowano domyślne pole identyfikatora, które będzie kombinacją RowID.
Dostęp do danych
Masz do wyboru wiele narzędzi wizualnych, aby uzyskać dostęp do zindeksowanych obrazów. HUE i Solr GUI to bardzo dobre opcje. HBase umożliwia również wiele technik dostępu, nie tylko z GUI, ale także przez powłokę HBase, API, a nawet proste techniki skryptowe.
Integracja z Solr daje Ci dużą elastyczność, a także może zapewnić bardzo proste i zaawansowane opcje wyszukiwania Twoich danych. Na przykład skonfigurowanie pliku schema.xml Solr w taki sposób, aby wszystkie pola w obiekcie e-mail były przechowywane w Solr, umożliwia użytkownikom dostęp do pełnej treści wiadomości poprzez proste wyszukiwanie, z kompromisem między przestrzenią dyskową a złożonością obliczeniową. Alternatywnie możesz skonfigurować Solr tak, aby przechowywał tylko ograniczoną liczbę pól, takich jak id. Dzięki tym elementom użytkownicy mogą szybko przeszukać Solr i pobrać rowID, który z kolei może zostać wykorzystany do pobrania poszczególnych pól lub całego obrazu z samego HBase.
Powyższy przykład przechowuje tylko rowID w Solr, ale indeksy wszystkich pól wyodrębnionych z obrazu. Wyszukiwanie Solr w tym scenariuszu pobiera identyfikatory wierszy HBase, które można następnie wykorzystać do zapytania HBase. Ten rodzaj konfiguracji jest idealny dla Solr, ponieważ utrzymuje niskie koszty przechowywania i w pełni wykorzystuje możliwości indeksowania Solr.
Przykładowe zapytania
Poniżej kilka przykładowych zapytań, które można wykonać z aplikacji do Solr. Pomysł polega na tym, że klient początkowo będzie odpytywał indeksy Solr, zwracając rowID z HBase. Następnie zapytaj HBase o pozostałe pola i/lub oryginalny surowy obraz.
- Proszę podać wszystkie dokumenty, które zostały złożone między następującymi datami:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=received:[2010-01-06T23:59:59.999Z TO 2010-02-06T23:59:59.999Z]
- Daj mi dokumenty, które zostały zgłoszone pod nazwą prawną mobilnych systemów rentgenowskich:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=reg_name:Mobile x-ray system
- Proszę podać wszystkie dokumenty złożone od chińskich producentów:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=submitter_info:*China*
Identyfikatory z dokumentów Solr to identyfikatory wierszy w HBase; druga część zapytania będzie skierowana do HBase w celu wyodrębnienia danych (w tym surowego pliku PDF, jeśli jest to wymagane).
Dostęp przez HUE
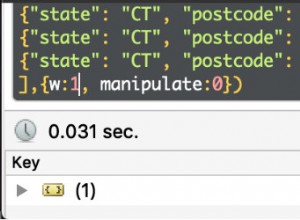
Wgrane dane możemy przeglądać za pomocą przeglądarki HBase w HUE. Jedną wielką zaletą HUE jest to, że może wykrywać pliki binarne dla PDF i renderować je po kliknięciu.
Poniżej znajduje się migawka widoku przeanalizowanych pól w wierszach HBase, a także renderowany widok jednego z obiektów PDF przechowywanych jako MOB w rodzinie kolumn obj.
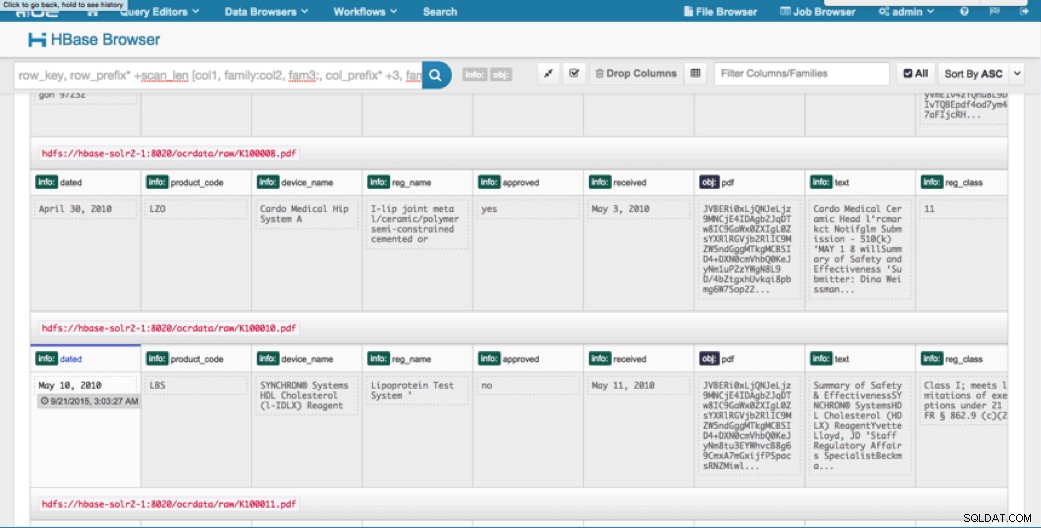
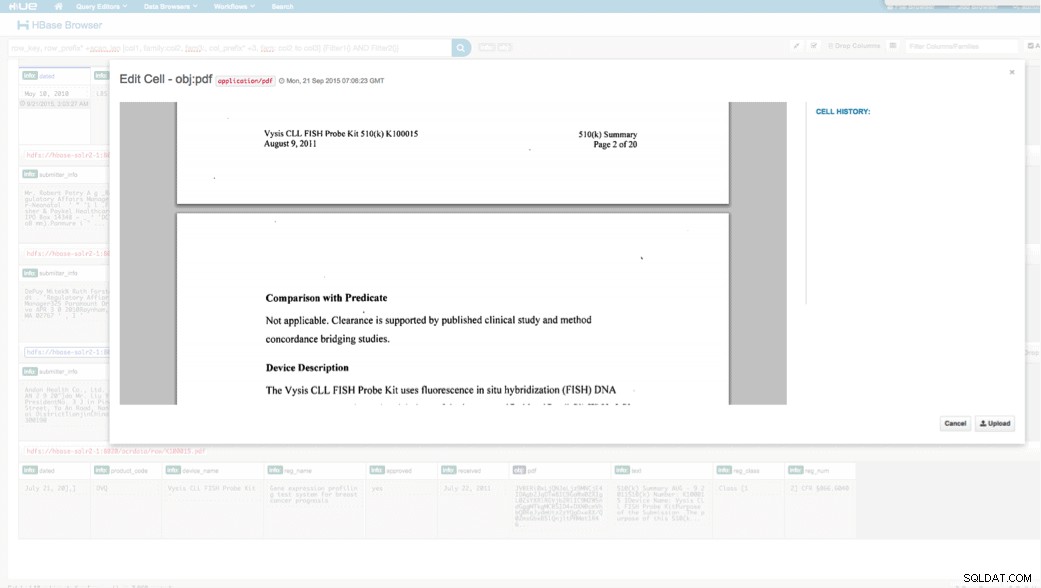
Wniosek
W tym poście pokazaliśmy, jak używać standardowych technologii open source do wykonywania OCR na zeskanowanych dokumentach za pomocą skalowalnego programu Spark, zapisywania w HBase w celu szybkiego wyszukiwania i indeksowania wyodrębnionych informacji w Solr. Powinno być oczywiste, że:
- Biorąc pod uwagę format specyfikacji wiadomości, możemy wyodrębnić pola i pary wartości i umożliwić ich przeszukiwanie przez Solr.
- Te pola z danych mogą spełnić wymagania IDMP dotyczące uczynienia starszych danych elektronicznymi, które wejdą w życie w przyszłym roku.
- Pola, a także nieprzetworzone obrazy mogą być utrwalane w HBase i dostępne za pośrednictwem standardowych interfejsów API.
Jeśli potrzebujesz przetworzyć zeskanowane dokumenty i połączyć dane z różnymi innymi źródłami w swoim przedsiębiorstwie, rozważ użycie kombinacji Spark, HBase, Solr wraz z Tesseract i Leptonica. Może to zaoszczędzić znaczną ilość czasu i pieniędzy!
Jeff Shmain jest starszym architektem rozwiązań w Cloudera. Ma ponad 16-letnie doświadczenie w branży finansowej z dużą znajomością handlu papierami wartościowymi, ryzyka i przepisów. W ciągu ostatnich kilku lat pracował nad różnymi wdrożeniami przypadków użycia w 8 z 10 największych banków inwestycyjnych na świecie.
Vartika Singh jest starszym konsultantem ds. rozwiązań w Cloudera. Ma ponad 12-letnie doświadczenie w stosowanym uczeniu maszynowym i tworzeniu oprogramowania.